ヒトを含む多くの生物種において、生殖細胞系譜は、精子及び卵子に分化し、それらが融合することで新しい個体をつくり、新しい世代に遺伝情報を伝える細胞系譜です。哺乳類の生殖細胞研究は、20世紀半ばの齧歯類を用いた初期胚培養系の確立・試験管内授精法の開発とともに発展し、その技術がヒトに応用され、1978年イギリスにおいて初の試験管ベビーが誕生するに至りました。現在、日本を含む先進国では、~30人に1人が試験管内授精を含む生殖補助医療により誕生し、世界で500万人以上がこれら技術により誕生しています。さらに、生殖細胞研究は、ES細胞の樹立・体細胞核移植によるクローンの作出・ゲノムインプリント*の発見等をもたらし、これらの研究は、転写因子により体細胞をリプログラミングするiPS細胞の樹立につながりました。生殖細胞研究は、基礎科学、医療、さらには生命哲学に大きな影響を及ぼしてきたと言えるでしょう。
我々の研究室では、生命の根源たる生殖細胞の発生機構を解明し、その過程を試験管内で再現することで、医学・生命科学さらには生命哲学の発展に貢献することを目的としています。また、マウス・サル・ヒト細胞を用いた研究を統合して推進することで、ゲノム・エピゲノム制御機構の進化に関する洞察を深めることを目指します(Saitou and Miyauchi, 2016)。
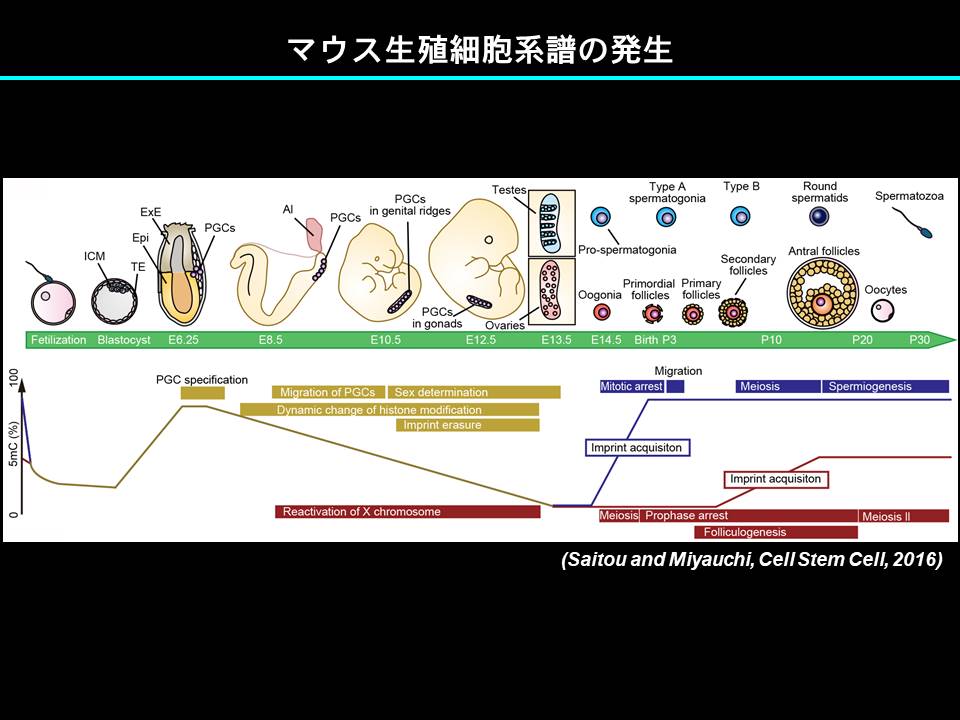
生殖細胞の発生過程
哺乳類における生殖細胞の発生機構の研究は、主にマウスをモデル生物として推進されてきました。生殖細胞系譜は、発生初期に形成される始原生殖細胞(primordial germ cells: PGCs)に由来します。PGCsは形成直後から、生殖巣(将来の精巣もしくは卵巣)に向け移動を開始し、発生中期に生殖巣に到達します。オス生殖巣(精巣)に到達した PGCs は gonocytes と呼ばれ、精原細胞・精原幹細胞(spermatogonia/spermatogonial stem cells: SSCs)へと分化し、生涯に渡って精子(spermatozoa)を産生し続ける準備を開始します。一方、メス生殖巣(卵巣)に到達したPGCsは卵原細胞(oogonia)と呼ばれ、減数分裂に入り、卵母細胞(oocytes)への分化・成熟を開始します。Gonocytes や卵原細胞が精原細胞や卵母細胞へとの分化するためには、生殖巣体細胞(セルトリ細胞もしくは顆粒膜細胞)との相互作用が必須です。
一方、生殖細胞は、運命決定後、移動中・生殖巣での増殖中に、そのエピゲノムを顕著にリプログラムします(エピゲノムリプログラミング)。例えば、形成直後のマウスPGCs(発生7日目頃)では、ゲノムワイドなCpG配列の約 70~80% がメチル化されていると考えられていますが、精原幹細胞や卵母細胞への分化を開始する直前のGonocytesや卵原細胞(発生13日目頃)では、そのレベルが 0% に近くなります。同時にヒストン修飾の大規模な変換もなされます。これに伴い、ゲノムインプリントが消去され、メスでは不活化していた1本のX染色体 * が再活性化されます。その結果、この時期の生殖細胞は、父母由来の染色体のエピゲノム状態が同一になる唯一の細胞であると考えられます。この後、精原細胞あるいは卵母細胞への分化に伴い、それぞれに特異的なエピゲノムが付与されます。
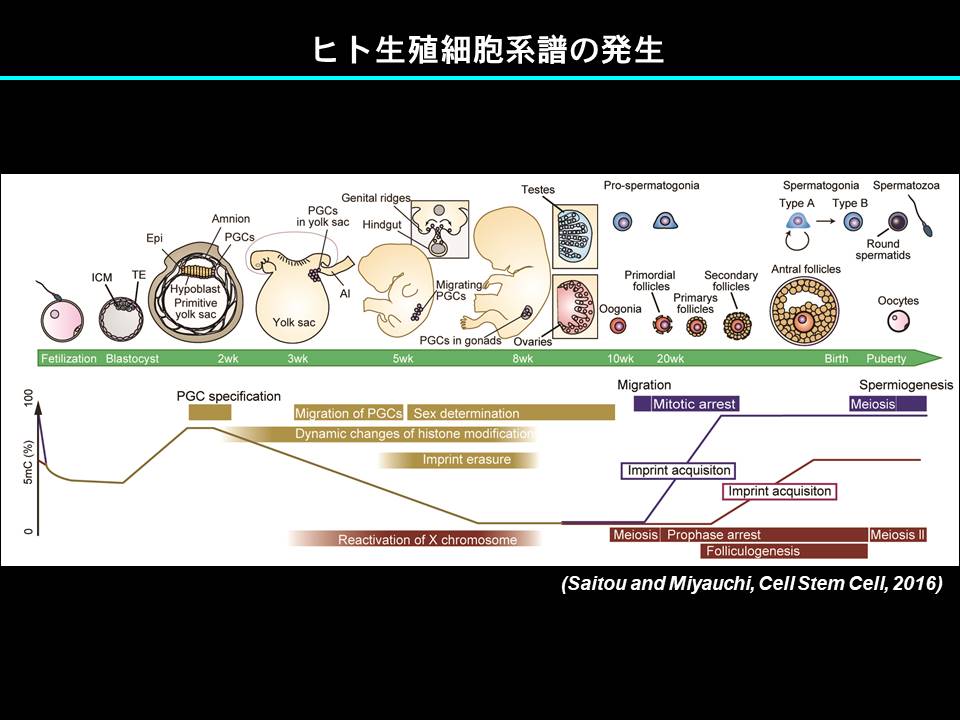
生殖細胞の発生は、ヒトでもほぼ同様の過程を経て進行します。ただし、ヒトではそれぞれの過程にマウスと比べてかなり長い時間を要します。ヒトPGCsが形成されるのは発生2週目頃(詳細は現在も不明です。)で、それらが生殖巣に到達するのが発生6週目頃、この期間にエピゲノムリプログラミングが徐々に進行すると考えられます。その後、胎児精巣中で精原細胞への分化の開始が明らかになるのが発生12週目以降、胎児卵巣中で減数分裂を開始し卵母細胞への分化を開始するのが11週目以降となります。また、マウスと異なり、精原細胞や卵母細胞への分化開始のタイミングもかなりheterogeneityに富むようです。ヒトは40週前後で生まれますが、生後生殖細胞の分化は一旦10年以上の休止状態に入り、思春期以降、精原(幹)細胞からの精子形成、卵母細胞の減数分裂の再開と卵子の形成が始まります。マウスとヒトで生殖細胞の発生にこのような時間の差が生じるメカニズムは全く解明されていません。
これまでの研究
我々はマウスを用いて生殖細胞の発生機構やエピゲノムリプログラミングの分子機構を研究してきました。代表的な成果として、PGC形成過程に関与する分子プログラムの提唱(Saitou et al., 2002)、PGC形成に必須な転写制御因子BLIMP1及びPRDM14の同定(Ohinata et al., 2005; Yamaji et al., 2008)、単一細胞cDNAマイクロアレイ法の開発とその応用(Kurimoto et al., 2006; Kurimoto et al., 2008)、PGCにおけるエピゲノムリプログラミング動態の提唱(Kagiwada et al., 2013; Seki et al., 2007)、PGC形成過程に関与するシグナル原理の解明(Ohinata et al., 2009)、等があります。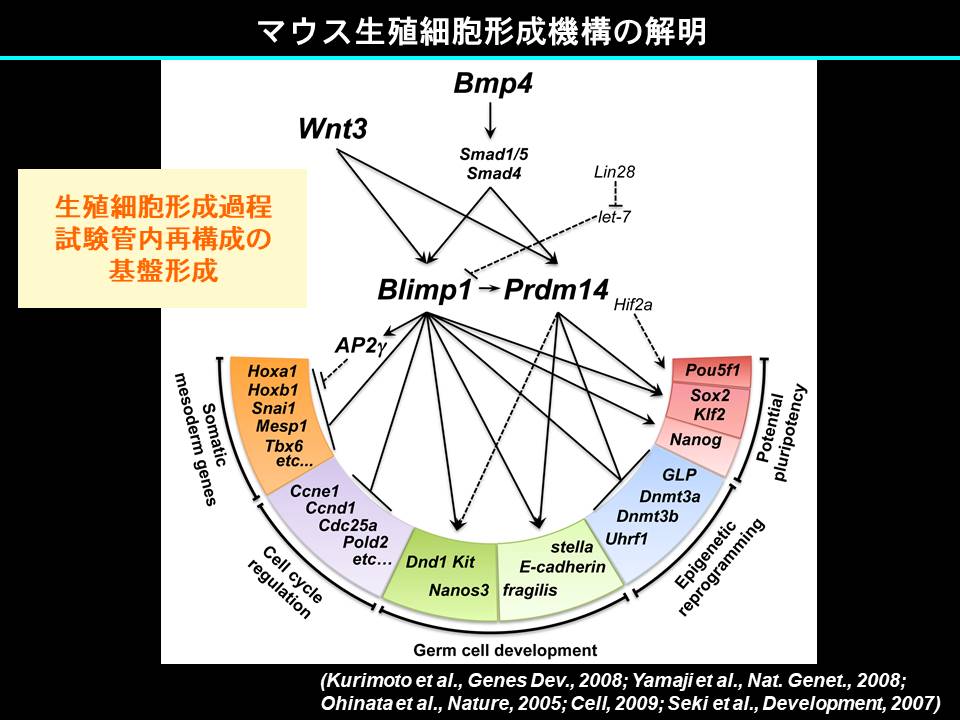
これら成果に基づき、我々は、培養ディッシュ上で、マウスES細胞/iPS細胞から、精子、卵子、さらには健常な産仔に貢献する能力を有する始原生殖細胞様細胞(primordial germ cell-like cells: PGCLCs)を誘導する技術を開発しました (Hayashi et al., 2012; Hayashi et al., 2011)。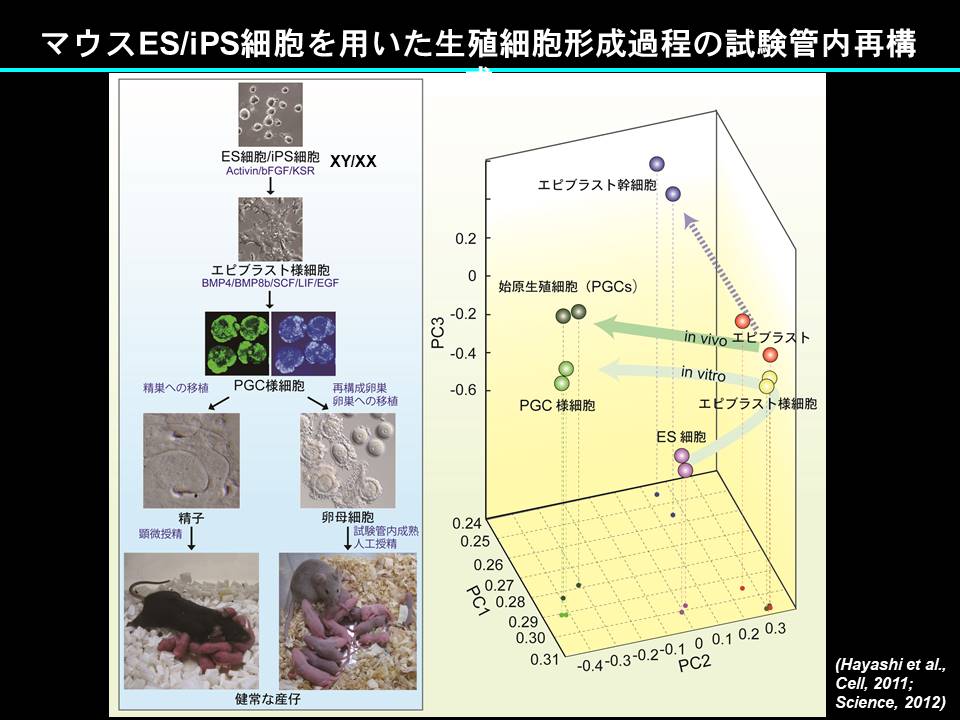
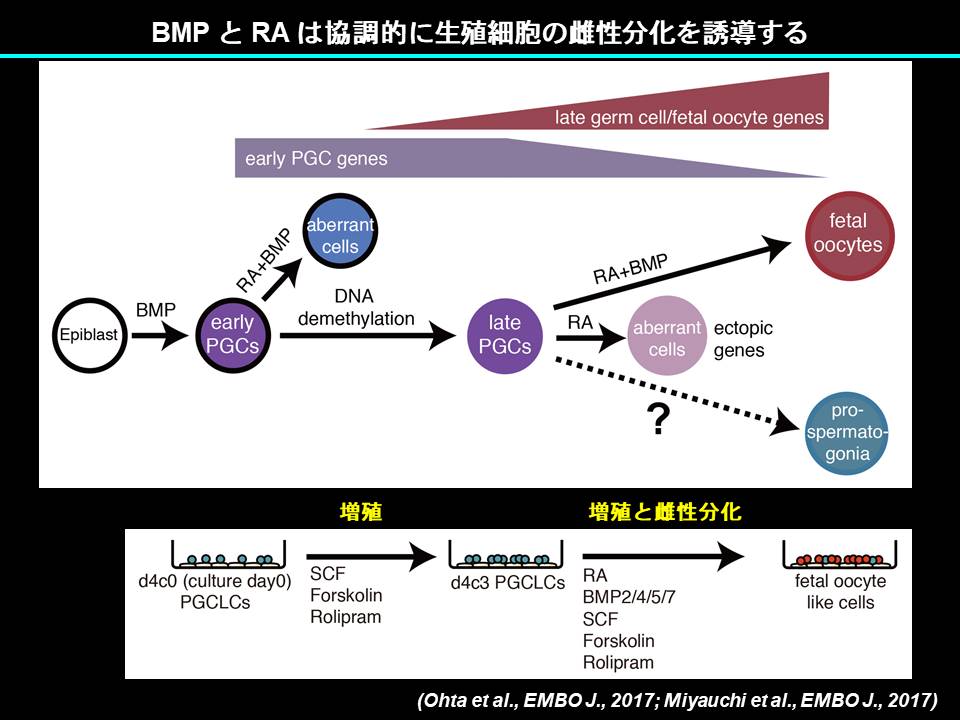
本技術を用いて、PGCsの誘導に十分な転写因子の同定(Nakaki et al., 2013)、PGC形成に関する中胚葉因子Tの機能解明(Aramaki et al., 2013)、エピゲノムリプログラミングの本態の解明(Kurimoto et al., 2015; Yamaji et al., 2013) (Shirane et al., 2016)に成功しました。さらに最近、PGCLCsとオスもしくはメス生殖巣体細胞を凝集させ適切な条件で培養することで(再構成精巣もしくは再構成卵巣)、PGCLCsをそれぞれ精原幹細胞様細胞もしくは成熟卵母細胞(本研究室から九州大学に独立した林克彦教授との共同研究)に分化させ、それぞれから産仔を得ることに成功しました (Hikabe et al., 2016; Ishikura et al., 2016)。これらの成果は哺乳類の生殖細胞研究を大きく発展させた成果として高く評価されています。実際に、これらの成果を基盤として、PGC(LC)sの増殖法の開発とそれに伴うエピゲノムリプログラミングの誘導 (Ohta et al., 2017)、生殖細胞の雌性化機構の解明 (Miyauchi et al., 2017)性染色体異常に起因する不妊マウスからの産仔の作製 (Hirota et al., 2017)、などに成功しました。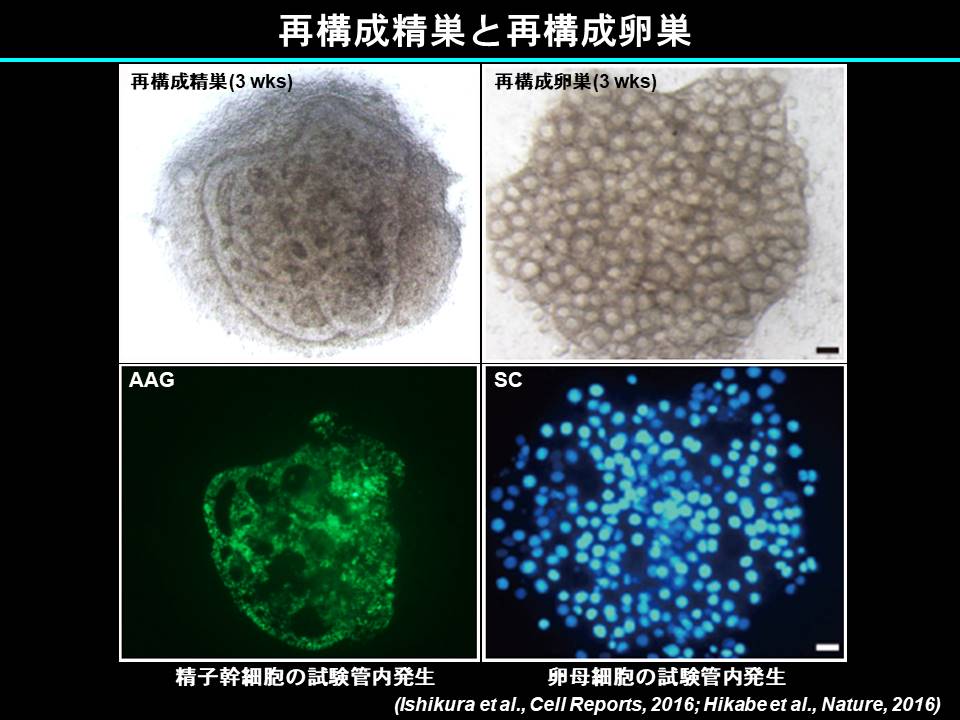
我々は、これら成果を基盤に、ヒトiPS細胞からヒトPGCLCsを誘導出来ることを示しました(Sasaki et al., 2015)。 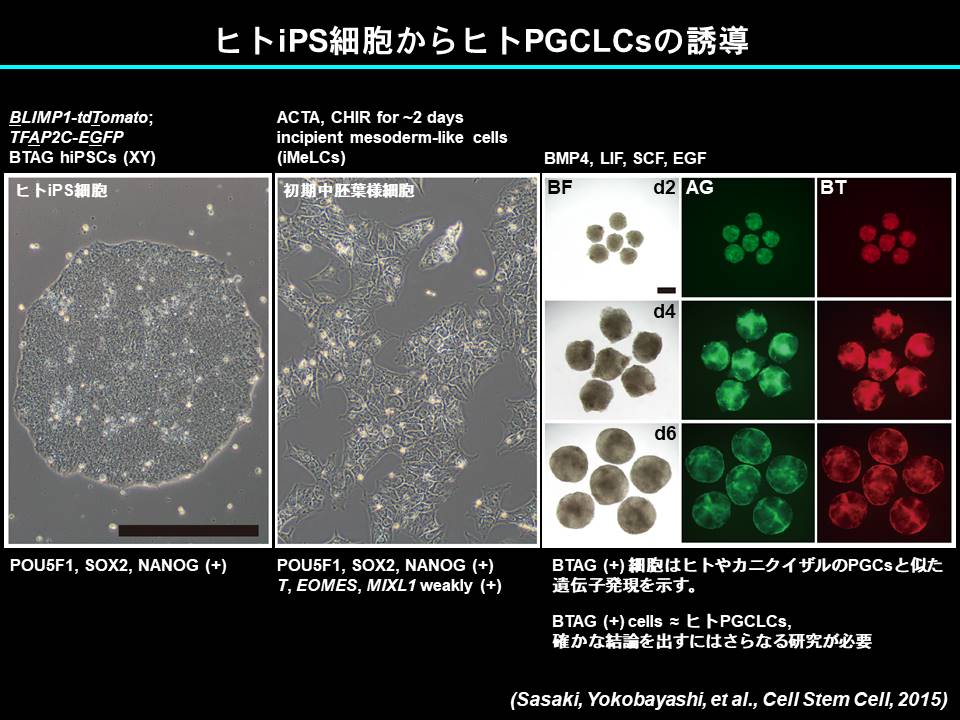
さらに、我々は、ヒト生殖細胞の試験管内誘導技術を発展させるため、実験動物として使用しうる霊長類の中でヒトに最も近縁のカニクイザルを用いた研究を推進しています。我々は、ヒトiPS細胞は着床後1週間程を経たカニクイザルの多能性細胞(エピブラスト)と似た特性を有することを証明し、マウス・サル・ヒトにおける多能性スペクトラムの発生座標を規定することに成功しました (Nakamura et al., 2016; Nakamura et al., 2015) 。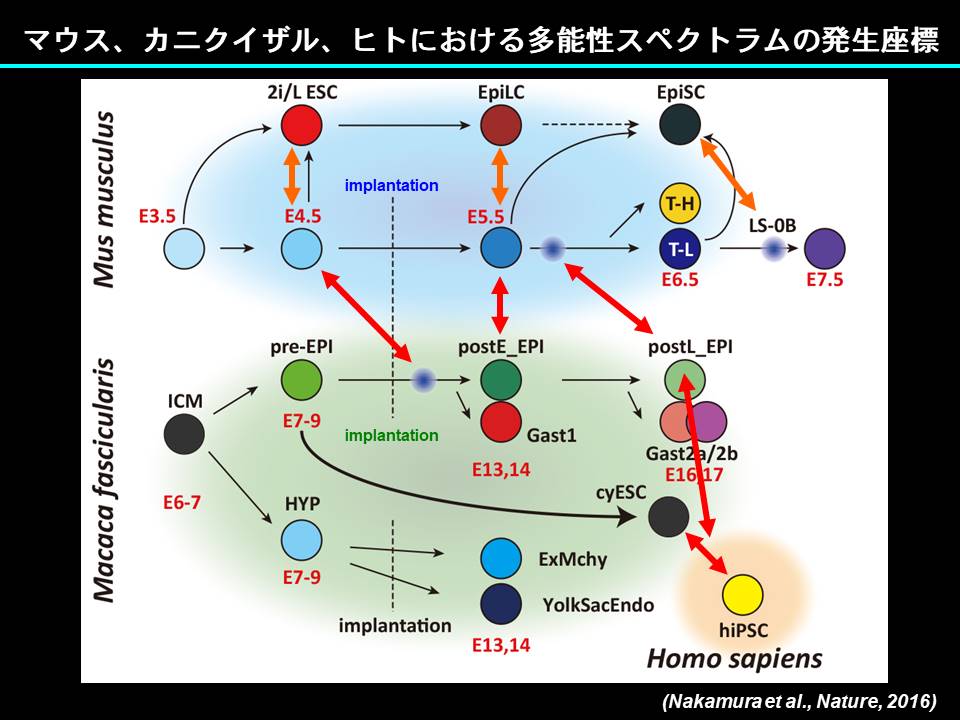
またカニクイザルの生殖細胞系譜の発生過程を詳細に解析することで、カニクイザルのPGCsが初期羊膜を起源とすることを見出しました (Sasaki et al., 2016)。ヒトを含む霊長類では、着床後エピブラストからまず羊膜が分化します。我々の発見は、ヒトPGCsも羊膜を起源とすることを強く示唆しています。またカニクイザルPGCsとヒトPGCLCsの遺伝子発現を比較することで、ヒトPGCLCsが形成直後のPGCsの特性を有することも証明しました (Sasaki et al., 2016; Sasaki et al., 2015)。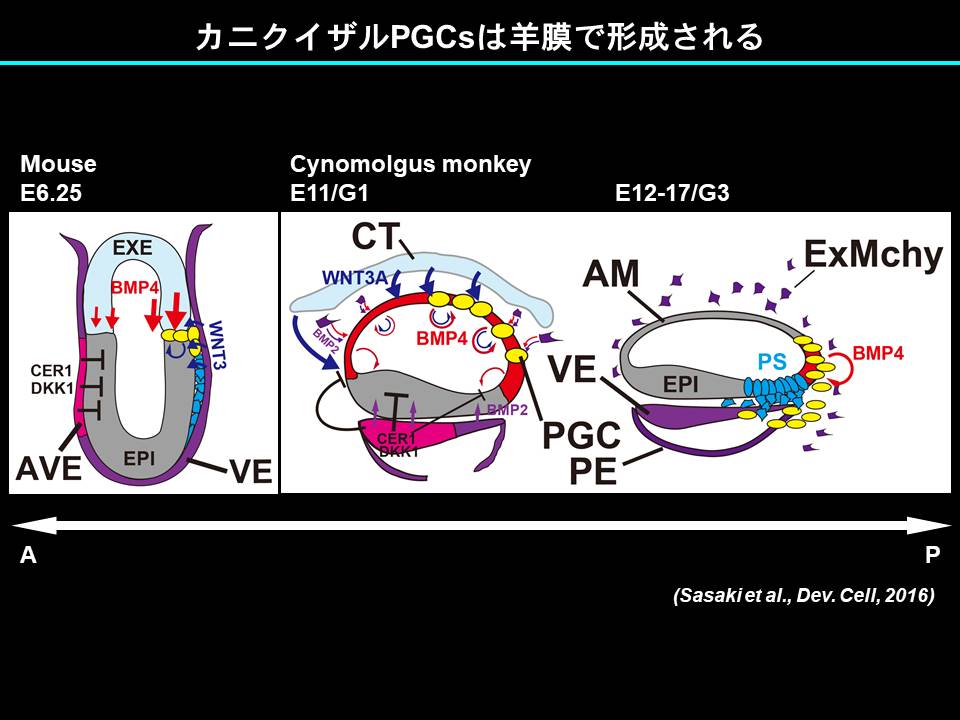
さらに、ヒトiPS細胞からのヒトPGCLCs誘導機構を詳細に解析することで、生殖細胞の形成に関与する鍵となる転写制御因子・その機能の階層性がヒトとマウスでは異なることを見出しました。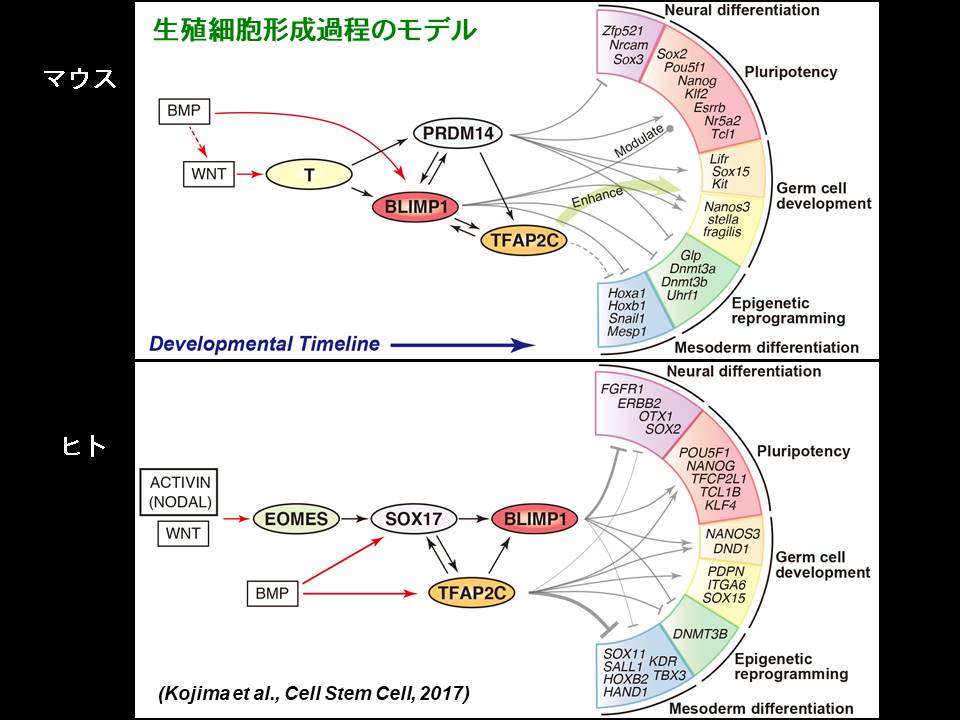
現在、これらの研究をさらに発展させるため、下記の研究を推進しています。
研究テーマ
マウス生殖細胞の発生機構の解明とその試験管内再構成
これまでの研究から、マウスPGCLCsは、オスもしくはメス生殖巣体細胞と凝集培養(再構成精巣もしくは再構成卵巣)することで、それぞれ精原幹細胞様細胞もしくは成熟卵母細胞に分化することがわかっています。一方で、誘導されたこれらの細胞はエピゲノム制御異常に起因する様々な異常を呈し、試験管内再構成系には改善の余地があることも明らかです。
本分野では、マウスPGC(LC)sの増殖機構の解明とPGC(LC)sの増殖法の開発・改善、規定条件下でPGC(LC)sの雌雄分化(精原幹細胞 vs 卵母細胞)を誘導する方法を開発しそのメカニズムを解明、再構成精巣法を改善しPGCLCs を高い効率で精原幹細胞様細胞に誘導する方法論を確立しそのメカニズムを解明、精原幹細胞様細胞に由来するエピゲノム異常の次世代への伝達の有無の解明、などを目指します。
カニクイザル生殖細胞の発生機構の解明とその試験管内再構成
これまでの研究で、着床前・後発生期におけるカニクイザル多能性細胞系譜の詳細な遺伝子発現プロファイル及び生殖細胞形成機構を解明しました。
本分野では、これら情報を基盤に、カニクイザル多能性幹細胞の培養法の改善、カニクイザル多能性幹細胞を起点としたカニクイザル生殖細胞発生過程の試験管内再構成、カニクイザル初期胚におけるX染色体の動態解明、などを目指します。本研究は滋賀医科大学動物生命科学研究センターと密接な連携のもと推進します。
ヒト生殖細胞の発生機構の解明とその試験管内再構成
これまでの研究で、ヒトiPS細胞からヒトPGCLCsを誘導することに成功しました。
本分野では、本成果と、マウス・カニクイザルを用いて得られた研究成果を基盤に、ヒトiPS細胞を起点としたヒト生殖細胞発生過程の再構成系の発展とその評価系の構築を目的とします。具体的には、多数のヒトiPS細胞株の特性とヒトPGCLC誘導効率の検証、XXヒトiPS細胞におけるX染色体の特性解明、ヒトPGCLCsを後期PGC様細胞に分化させ、その過程におけるエピゲノム動態の解明、ヒトPGCLCsの雌雄分化誘導法の開発(精原細胞や卵母細胞への分化誘導)とそのメカニズムの解明、などを目指します。本研究は京都大学iPS細胞研究所と連携して行います。
ヒト生殖巣体細胞発生機構の解明とその試験管内再構成
ヒトPGCLCsを精原幹細胞や成熟卵母細胞に最終分化させるには、ヒト生殖巣体細胞との凝集培養が必要となる可能性が高いと考えられます。しかしながら、適切な発生時期のヒト生殖巣体細胞の恒常的な入手は、技術的にも倫理的にも容易ではありません。
本分野では、マウスにおける知見、近縁体細胞(embryonic kidney, etc.)の分化誘導法、カニクイザル胚を用いた知見を基に、ヒトiPS細胞からヒト生殖巣体細胞(Sertoli細胞や顆粒膜細胞)を誘導する方法を開発することを目指します。生殖巣体細胞の発生機構には未解明な点が多く残されており、着実に研究を進める必要がある研究分野であると考えられます。
*全能性:全能性とは、個体を構成するすべての細胞に分化し、個体を形成する能力のことを言います。我々ヒトを含む哺乳類において、全能性を有する細胞は、個体の出発点である受精卵とそれが分裂して形成される2細胞胚や4細胞胚を構成する細胞に限られます。その後の発生過程で、胚を構成する細胞から全能性は失われますが、次世代の精子や卵子の源となる始原生殖細胞(primordial germ cells: PGCs)の中で、全能性を再獲得するプログラムが開始されます。始原生殖細胞から精子や卵子へ、そして再び全能性を持つ受精卵へと、世代を超えて遺伝情報を継承する仕組みの中では、ゲノム機能を制御するゲノム上のさまざまな修飾(その総体をエピゲノムと呼びます)がダイナミックに変化することが知られています。全能性獲得に至るエピゲノム制御機構の理解は、医学・生命科学における基本命題の一つであるのみならず、細胞の医療応用の観点からも非常に重要です。
*体細胞と生殖細胞:ヒトを含む多細胞生物の体を構成する細胞は大きく体細胞と生殖細胞に分けられます。体細胞は、皮膚や神経、血液、筋肉、骨、腸や肝臓の細胞など、個体の生命活動の維持に必須な細胞です。一方生殖細胞は、精子や卵子など、個体の生命活動には必ずしも必要ではありませんが、次世代をつくるのに必須な細胞です。体細胞がその世代限りでその使命を終わらせるのに対し、生殖細胞は次の世代を、さらにはその次の世代をと、いわば果てしなく新しい生命を産み出し続けます。地球上には多種多様な生物が生命活動を繰り広げていますが、その多くは生殖細胞を介して種の存続を行い、種の進化も生殖細胞を介した遺伝子変異によって起こってきました。どうして生殖細胞だけがこうした能力を有するのでしょうか?生殖細胞の発生サイクルの特性を理解すれば、その答えが得られるのみならず、細胞の増殖や分化、老化といった現象の制御にもつながると期待されます。
*ゲノムインプリント:我々ヒトを含む哺乳類の細胞は、父方(精子)及び母方(卵子)から受け継いだそれぞれ一対の遺伝子を有しています(ヒトではタンパク質をコードする遺伝子の総数は約22,000)。ほとんどの遺伝子は、父方母方のアレルから同じように発現しますが、一部の遺伝子(百数十の遺伝子)は、父方もしくは母方のアレルのみから発現することが知られています。それらの遺伝子のことをインプリント遺伝子と言います。遺伝子のインプリントは、PGCsから精原幹細胞及び卵子が形成される過程で起こり、遺伝子発現を制御する領域のDNAメチル化やヒストン修飾により付与されます。インプリントは受精後の細胞で保たれますが、PGCsの中でのみ消去され、その後、上述のように性に応じたインプリントが付与されます。インプリントに異常が生じると様々な病態につながることが知られています。
*X 染色体不活化・再活性化:我々ヒトを含む多くの哺乳類の性は、性染色体により決定されます。通常メス(女性)はX染色体を2本有し(XX)、オス(男性)は1本のX染色体と1本のY染色体を有します(XY)。X染色体には1000を超える遺伝子が含まれていますが、Y染色体には、オス(男性)化及び精子形成過程に重要な少数の遺伝子が含まれるのみです。メス(女性)でX染色体が2本とも活性化された状態であると、メス(女性)とオス(男性)の間で1000を超える遺伝子の発現量に差が生じ、発生異常が生じます。そのため、メス(女性)の発生過程初期(PGC形成以前)に、1本のX染色体が不活化されます。この不活化されたX染色体は、PGCの発生過程で起こるエピゲノムリプログラミングに伴い再活性化されます。X染色体が2本とも活性化された状態にあることは卵子の形成過程に重要であると考えられています。
代表的な業績
- Aramaki, S., Hayashi, K., Kurimoto, K., Ohta, H., Yabuta, Y., Iwanari, H., Mochizuki, Y., Hamakubo, T., Kato, Y., Shirahige, K., et al. (2013). A mesodermal factor, T, specifies mouse germ cell fate by directly activating germline determinants. Dev Cell 27, 516-529.
- Hayashi, K., Ogushi, S., Kurimoto, K., Shimamoto, S., Ohta, H., and Saitou, M. (2012). Offspring from oocytes derived from in vitro primordial germ cell-like cells in mice. Science 338, 971-975.
- Hayashi, K., Ohta, H., Kurimoto, K., Aramaki, S., and Saitou, M. (2011). Reconstitution of the mouse germ cell specification pathway in culture by pluripotent stem cells. Cell 146, 519-532.
- Hikabe, O., Hamazaki, N., Nagamatsu, G., Obata, Y., Hirao, Y., Hamada, N., Shimamoto, S., Imamura, T., Nakashima, K., Saitou, M., et al. (2016). Reconstitution in vitro of the entire cycle of the mouse female germ line. Nature 539, 299-303.
- Hirota, T., Ohta, H., Powell, B.E., Mahadevaiah, S.K., Ojarikre, O.A., Saitou, M., and Turner, J.M.A. (2017). Fertile offspring from sterile sex chromosome trisomic mice. Science 357, 932-935.
- Ishikura, Y., Yabuta, Y., Ohta, H., Hayashi, K., Nakamura, T., Okamoto, I., Yamamoto, T., Kurimoto, K., Shirane, K., Sasaki, H., et al. (2016). In Vitro Derivation and Propagation of Spermatogonial Stem Cell Activity from Mouse Pluripotent Stem Cells. Cell Reports 17, 2789-2804.
- Kagiwada, S., Kurimoto, K., Hirota, T., Yamaji, M., and Saitou, M. (2013). Replication-coupled passive DNA demethylation for the erasure of genome imprints in mice. EMBO J 32, 340-353.
- Kurimoto, K., Yabuta, Y., Hayashi, K., Ohta, H., Kiyonari, H., Mitani, T., Moritoki, Y., Kohri, K., Kimura, H., Yamamoto, T., et al. (2015). Quantitative Dynamics of Chromatin Remodeling during Germ Cell Specification from Mouse Embryonic Stem Cells. Cell Stem Cell 16, 517-532.
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Ono, Y., Uno, K.D., Yamada, R.G., Ueda, H.R., and Saitou, M. (2006). An improved single-cell cDNA amplification method for efficient high-density oligonucleotide microarray analysis. Nucleic Acids Res 34, e42.
- Kurimoto, K., Yabuta, Y., Ohinata, Y., Shigeta, M., Yamanaka, K., and Saitou, M. (2008). Complex genome-wide transcription dynamics orchestrated by Blimp1 for the specification of the germ cell lineage in mice. Genes Dev 22, 1617-1635.
- Miyauchi, H., Ohta, H., Nagaoka, S., Nakaki, F., Sasaki, K., Hayashi, K., Yabuta, Y., Nakamura, T., Yamamoto, T., and Saitou, M. (2017).Bone morphogenetic protein and retinoic acid synergistically specify female germ-cell fate in mice. EMBO J 36,3100-3119.
- Nakaki, F., Hayashi, K., Ohta, H., Kurimoto, K., Yabuta, Y., and Saitou, M. (2013). Induction of mouse germ-cell fate by transcription factors in vitro. Nature 501, 222-226.
- Nakamura, T., Okamoto, I., Sasaki, K., Yabuta, Y., Iwatani, C., Tsuchiya, H., Seita, Y., Nakamura, S., Yamamoto, T., and Saitou, M. (2016). A developmental coordinate of pluripotency among mice, monkeys and humans. Nature 537, 57-62.
- Nakamura, T., Yabuta, Y., Okamoto, I., Aramaki, S., Yokobayashi, S., Kurimoto, K., Sekiguchi, K., Nakagawa, M., Yamamoto, T., and Saitou, M. (2015). SC3-seq: a method for highly parallel and quantitative measurement of single-cell gene expression. Nucleic Acids Res 43, e60.
- Ohinata, Y., Ohta, H., Shigeta, M., Yamanaka, K., Wakayama, T., and Saitou, M. (2009). A signaling principle for the specification of the germ cell lineage in mice. Cell 137, 571-584.
- Ohinata, Y., Payer, B., O'Carroll, D., Ancelin, K., Ono, Y., Sano, M., Barton, S.C., Obukhanych, T., Nussenzweig, M., Tarakhovsky, A., et al. (2005). Blimp1 is a critical determinant of the germ cell lineage in mice. Nature 436, 207-213.
- Ohta, H., Kurimoto, K., Okamoto, I., Nakamura, T., Yabuta, Y., Miyauchi, H., Yamamoto, T., Okuno, Y., Hagiwara, M., Shirane, K.,et al. (2017). In vitro expansion of mouse primordial germ cell-like cells recapitulates an epigenetic blank slate. EMBO J36, 1888-1907.
- Saitou, M., Barton, S.C., and Surani, M.A. (2002). A molecular programme for the specification of germ cell fate in mice. Nature 418, 293-300.
- Saitou, M., and Miyauchi, H. (2016). Gametogenesis from Pluripotent Stem Cells. Cell Stem Cell 18, 721-735.
- Sasaki, K., Nakamura, T., Okamoto, I., Yabuta, Y., Iwatani, C., Tsuchiya, H., Seita, Y., Nakamura, S., Shiraki, N., Takakuwa, T., et al. (2016). The Germ Cell Fate of Cynomolgus Monkeys Is Specified in the Nascent Amnion. Dev Cell 39, 169-185.
- Sasaki, K., Yokobayashi, S., Nakamura, T., Okamoto, I., Yabuta, Y., Kurimoto, K., Ohta, H., Moritoki, Y., Iwatani, C., Tsuchiya, H., et al. (2015). Robust In Vitro Induction of Human Germ Cell Fate from Pluripotent Stem Cells. Cell Stem Cell 17, 178-194.
- Seki, Y., Yamaji, M., Yabuta, Y., Sano, M., Shigeta, M., Matsui, Y., Saga, Y., Tachibana, M., Shinkai, Y., and Saitou, M. (2007). Cellular dynamics associated with the genome-wide epigenetic reprogramming in migrating primordial germ cells in mice. Development 134, 2627-2638.
- Shirane, K., Kurimoto, K., Yabuta, Y., Yamaji, M., Satoh, J., Ito, S., Watanabe, A., Hayashi, K., Saitou, M., and Sasaki, H. (2016). Global Landscape and Regulatory Principles of DNA Methylation Reprogramming for Germ Cell Specification by Mouse Pluripotent Stem Cells. Dev Cell 39, 87-103.
- Yamaji, M., Seki, Y., Kurimoto, K., Yabuta, Y., Yuasa, M., Shigeta, M., Yamanaka, K., Ohinata, Y., and Saitou, M. (2008). Critical function of Prdm14 for the establishment of the germ cell lineage in mice. Nat Genet 40, 1016-1022.
- Yamaji, M., Ueda, J., Hayashi, K., Ohta, H., Yabuta, Y., Kurimoto, K., Nakato, R., Yamada, Y., Shirahige, K., and Saitou, M. (2013). PRDM14 ensures naive pluripotency through dual regulation of signaling and epigenetic pathways in mouse embryonic stem cells. Cell Stem Cell 12, 368-382.
